La genética es una rama de la biología que ha crecido mucho últimamente, la tecnología nos permite ahora conocer los genes de prácticamente cualquier ser vivo. Pero, ¿de qué nos sirve?
Seguramente la respuesta es que nos sirve para entender algunas enfermedades y ya, sin embargo, es una sola de las múltiples posibilidades. En realidad, al tener acceso a los genes de cualquier ser vivo se abre el código de la vida, lo que significa entender los procesos biológicos en sí mismos y un poco del pasado de los seres vivos, “la historia contada por los genes”; además de visualizar algo de su futuro con “la frecuencia de genes en las poblaciones”.
Es cierto que la humanidad ya conoce la secuencia completa del ADN (ácido desoxirribonucleico) de nuestra especie. Esta molécula porta la información hereditaria y la información ancestral de la historia humana.
Al decir “la secuencia completa” me refiero a que el ADN está compuesto por cuatro tipos de moléculas más pequeñas, las bases nitrogenadas: adenina (A), timina (T), guanina (G) y citocina (C), y su secuencia (ejemplo: AATTCAT o CCATGC) ya es conocida para los 3 mil 200 millones de pares de bases que componen todo el ADN del ser humano.
De forma correcta, podemos decir que todo el ADN característico de una especie es su genoma (Figura 1), el genoma entonces es toda la secuencia de genes y otras secuencias que no son genes.
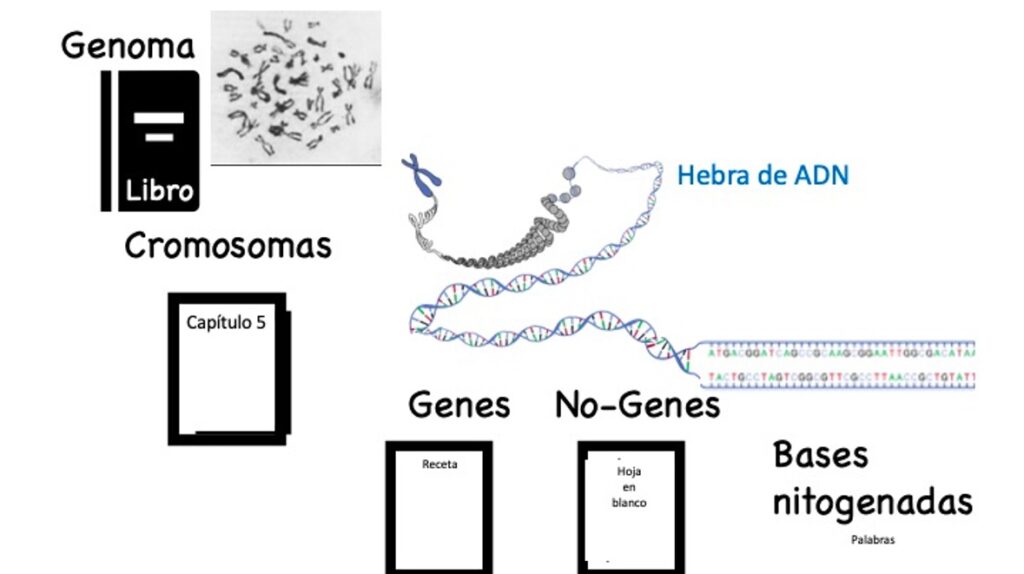
Entonces, tenemos genes que son los que dictan las características de los organismos y tenemos secuencias no-genes. Estas últimas, ¿qué hacen o para qué sirven? Pueden tener muchas funciones, como ser “instrucciones” que dicen qué gen se expresa y cuál no; pueden ser “estructurales”, o sea sitios de ensamble para componentes celulares (como el aparato mitótico) o secuencias de “protección”(por ejemplo, los telómeros, que es la parte final del cromosoma, que se pierde cada que la célula se divide); y también pueden ser sitios entre genes o intra-genes (como los intrones), que son secuencias que simplemente parecen no tener una función, pero están ahí y a veces repetidos en algún número de copias.
Ahora, ¿todas estas secuencias, codificantes y no codificantes, nos sirven para entender a los seres vivos? ¿Cómo?
Vamos a platicar de algunos casos muy concretos para ejemplificar las posibilidades de “uso” del entendimiento de los procesos biológicos. Pero primero necesitamos saber que todos los seres vivos de este planeta (bacterias, hongos, plantas, animales, seres humanos, e incluso seres no tan vivos como los virus) usan las mismas bases nitrogenadas: adenina, timina, guanina y citocina, ordenadas en una molécula de mayor tamaño para pasar la información de sus características a su descendencia. Estas moléculas son el ADN y el ARN (ácido desoxirribonucleico), que en lugar de poseer timina tiene uracilo (otra base nitrogenada).
Por consiguiente, todos los seres portan información hereditaria y evolutiva de la misma manera. Entender el genoma de una especie inmediatamente nos sirve para hacer inferencias en otras especies.
Los ejemplos concretos son:
Enfermedades humanas
Mencionamos que nos sirve conocer la secuencia del genoma humano porque podemos identificar las enfermedades que son consecuencia de la genética, es decir, qué secuencia codificante (que se expresa) o reguladora tiene algún cambio en la secuencia de bases que está provocando una enfermedad.
Terapias y cura para las enfermedades
Entendemos que algunas regiones de la molécula del ADN están silenciadas, por ejemplo, la segunda copia del cromosoma X en hembras (Wutz, 2011); estos mecanismos de silenciamiento podrían ser usados para silenciar otros cromosomas que “estén de más”, como el caso del cromosoma 21 en el síndrome de Down.
Genes útiles
Los distintos genomas de las especies “guardan” información en forma de genes. Las ranas, por ejemplo, pueden tener genes para toxinas (You et al., 2009), los hongos pueden poseer genes anti-hongos (Liu et al., 2006), los ajolotes genes asociados a la regeneración de tejidos (Barradas, 2020), etc. Tantos organismos representan un recurso invaluable de información que fue almacenada y perfeccionada a través de millones de años.
Relaciones entre organismos
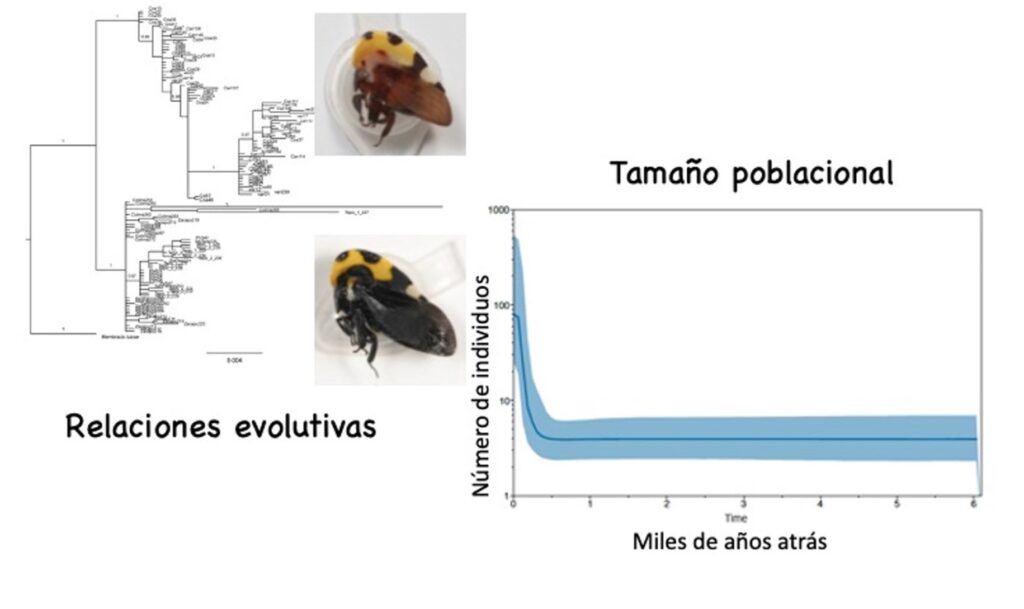
Dos organismos que se parecen, así como los hermanos, comparten ADN, es decir, secuencias idénticas de nucleótidos. Entre más alejados estén, como primos, irán compartiendo menos. Así es posible por medio del ADN conocer cómo se encuentran relacionados los seres vivos en el planeta.
Procesos demográficos históricos
Mencionamos que hay secuencias en el genoma que no son genes. Al no expresarse, la selección natural no puede favorecer que se mantengan o eliminen en las poblaciones, por lo que la gran o poca diversidad que encontremos en estas secuencias en los organismos de una especie nos puede “decir” qué tantos individuos hay o ha habido a lo largo del tiempo, teniendo así la historia de las poblaciones (Emerson et al., 2001).
Procesos poblacionales frecuentes
Pasar la molécula de ADN de una generación a la siguiente provoca que la generación de hijos se vea cómo sus padres; al tomar en cuenta qué individuos se reproducen y cuáles no, podemos modelar la dinámica de cambio evolutivo y saber cómo será la población en unas pocas generaciones al futuro (Nosil et al. 2018).
Conservación y sustentabilidad de la vida
Literalmente, cada ser vivo es único en la secuencia de bases que poseen, a excepción de los que se reproducen asexualmente, por ejemplo, las plantas, que al reproducirse de forma vegetativa siguen conservando la misma secuencia de ADN de una planta a otra. Es por lo que en los esfuerzos de reforestación se debe procurar que los individuos sembrados sean variables genéticamente (sus secuencias de nucleótidos sean distintas), que no sean clones, porque pueden ser fácilmente atacados por plagas o patógenos y no sobrevivirán a largo plazo (ejemplo, Salas-Leiva et al., 2009).
Estos son algunos de los usos que puede tener el conocimiento del ADN a tan sólo 69 años de la descripción de su estructura (Watson & Crick, 1953).
La doctora Marisol de la Mora Curiel es profesora de la Escuela Nacional de Estudios Superiores, Unidad Juriquilla, de la Universidad Nacional Autónoma de México.
AQUI PUEDES LEER TODAS LAS ENTREGAS DE “DESDE LA UNAM”, LA COLUMNA DE LA UNAM, CAMPUS JURIQUILLA, PARA LALUPA.MX
https://lalupa.mx/category/aula-magna/desde-la-unam/
Literatura citada
Barradas Chacón, C. D. (2020). Identification and characterization of microRNAs involved in limb regeneration of Ambystoma mexicanum= Identificación y caracterización de microRNAs involucrados en la regeneraciónde extremidad de Ambystoma mexicanum (Master’s thesis, Tesis (MC)–Centro de Investigación y de Estudios Avanzados del IPN Unidad Irapuato. Laboratorio Nacional de Genómica para la Biodiversidad).
Emerson, B. C., Paradis, E., & Thébaud, C. (2001). Revealing the demographic histories of species using DNA sequences. Trends in Ecology & Evolution, 16(12), 707-716.
Liu, M., Healy, M. D., Dougherty, B. A., Esposito, K. M., Maurice, T. C., Mazzucco, C. E., … & Wang, Y. K. (2006). Conserved fungal genes as potential targets for broad-spectrum antifungal drug discovery. Eukaryotic Cell, 5(4), 638-649.
Nosil, P., Villoutreix, R., de Carvalho, C. F., Farkas, T. E., Soria-Carrasco, V., Feder, J. L., … & Gompert, Z. (2018). Natural selection and the predictability of evolution in Timema stick insects. Science, 359(6377), 765-770.
Salas-Leiva, D. E., Mayor-Durán, V. M., & Toro-Perea, N. (2009). Genetic diversity of black mangrove (Avicennia germinans) in natural and reforested areas of Salamanca Island Parkway, Colombian Caribbean. Hydrobiologia, 620(1), 17-24.
Watson, J. D., & Crick, F. H. (1953, January). The structure of DNA. In Cold Spring Harbor symposia on quantitative biology (Vol. 18, pp. 123-131). Cold Spring Harbor Laboratory Press.
Wutz, A. (2011). Gene silencing in X-chromosome inactivation: advances in understanding facultative heterochromatin formation. Nature Reviews Genetics, 12(8), 542-553.
You, D., Hong, J., Rong, M., Yu, H., Liang, S., Ma, Y., … & Lai, R. (2009). The first gene-encoded amphibian neurotoxin. Journal of Biological Chemistry, 284(33), 22079-22086.
















