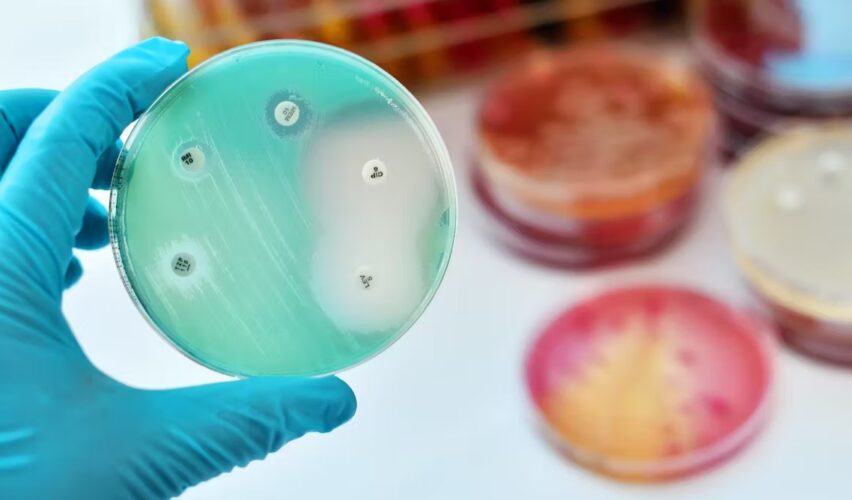
La resistencia a antibióticos es la capacidad de las bacterias de combatir la acción de uno o varios antibióticos, y estas bacterias pueden propagarse fácilmente por diversas vías (Figura 1). En este sentido, la resistencia a antibióticos es un problema de salud global creciente que representa una amenaza para la salud humana, la salud animal y el medio ambiente. En este problema, las aguas residuales contribuyen a la diseminación de genes de resistencia y bacterias resistentes en el medio ambiente. En términos muy simples, estos genes de resistencia contienen la información genética necesaria para que las bacterias puedan resistir el efecto adverso de los antibióticos.

Las plantas de tratamiento de aguas residuales (PTAR) juegan un papel crucial en la eliminación de antibióticos y genes de resistencia antes de la descarga del agua tratada al medio ambiente. Sin embargo, la efectividad de las tecnologías actuales de tratamiento de aguas residuales para eliminar la resistencia a antibióticos es limitada. Por ende, en la actualidad se están realizando esfuerzos para abordar el problema de la resistencia a antibióticos en las aguas residuales, incluido el desarrollo de nuevas tecnologías de tratamiento de aguas residuales y la implementación de políticas públicas para reducir el uso de antibióticos en la medicina humana, veterinaria y en la agricultura.
El monitoreo de la resistencia a antibióticos en las aguas residuales es esencial para comprender los niveles de agentes antimicrobianos y genes presentes en las aguas residuales, y para evaluar la eficacia de remoción de estos contaminantes en las tecnologías de tratamiento actuales. Dos métodos comunes utilizados para el monitoreo son la reacción cuantitativa en cadena de la polimerasa (qPCR) y la metagenómica, ambas técnicas basadas en el material genético presente en el agua residual.
La qPCR es una técnica de biología molecular que permite la detección y cuantificación rápida de secuencias de material genético específicas incluidos los genes en muestras ambientales. Sin embargo, la qPCR se limita a detectar solo genes específicos y no brinda información sobre la diversidad o composición de las comunidades microbianas en las aguas residuales. Por otro lado, la metagenómica, es una poderosa herramienta que permite analizar el material genético de todos los microorganismos presentes en una muestra. La metagenómica puede proporcionar una visión integral de las comunidades microbianas y los genes de resistencia presentes en las aguas residuales y su potencial para contribuir a la propagación de la resistencia a antibióticos. Sin embargo, la metagenómica es más compleja que la qPCR y requiere herramientas bioinformáticas avanzadas para el análisis de datos.
Recientemente, en la Unidad Académica del Instituto de Ingeniería del Campus Juriquilla de la UNAM, hemos implementado herramientas para el monitoreo de la resistencia a antibióticos en aguas residuales de la ciudad de Querétaro. Este procedimiento consistió en tomar muestras de agua residual, posterior a ello, se realizó la concentración del material genético. En este material se puede identificar una gran cantidad de genes de resistencia y bacterias. Posteriormente, se consideraron algunos de los genes de resistencia más predominantes y de relevancia clínica, para poder monitorearlos utilizando qPCR, lo cual nos permitió tener una cuantificación rápida y puntual de estos genes en el agua residual (Figura 2).

Los análisis de metagenómica relevaron la presencia de 238 diferentes tipos de GRA en el agua residual, donde los más abundantes fueron a aquellos que confieren resistencia a multidrogas, tetraciclinas, aminoglucósidos y betalactámicos. Además, las bacterias más predominantes fueron Enterobacteriales, estas bacterias han sido reportadas previamente como relevantes para la salud humana, ya que ocasionan infecciones urinarias, gastrointestinales y otros tipos de infecciones. Por otro lado, la qPCR nos permitió monitorear de manera más rápida y eficiente genes relacionados con resistencia a sulfonamidas, tetraciclinas y betalactámicos.
En conclusión, la combinación la qPCR y la metagenómica puede proporcionar una comprensión más completa de la resistencia antimicrobiana. La identificación y cuantificación de genes de resistencia a antibióticos en aguas residuales usando ambas herramientas, puede proporcionar información valiosa para el desarrollo de estrategias efectivas de tratamiento de aguas residuales y finalmente, para mitigar la propagación de resistencia a antibióticos al medio ambiente y la salud humana.
Información de interés
https://www.unep.org/es/resources/superbugs/environmental-action
Julián Osvaldo Ovis Sánchez es candidato a doctor en Ingeniería Ambiental por la UNAM. Julián Carrillo Reyes es Investigador del Instituto de Ingeniería UNAM, en la Unidad Académica Juriquilla.
AQUÍ PUEDES LEER TODAS LAS ENTREGAS DE “DESDE LA UNAM”, UNA COLUMNA DE UNAM, CAMPUS JURIQUILLA PARA LALUPA.MX
https://lalupa.mx/category/aula-magna/desde-la-unam/